De retour de Concarneau pour un “collaboration fest” orienté R, et plutôt ciblé pour des chercheurs en écologie. Au cours de cet événement, nous avons eu des formations diverses, du script R à la mise en production à la mise en conteneurs Docker sur la plateforme Galaxy, en passant par les applications Shiny. Nous avons aussi commencé un projet de transformation d’une série de scripts d’analyse vers le développement d’un package documenté, testé, versionné.
L’événement nommé “ecoinfo-fair : écoinformatique FAIR par la pratique” était organisé par le “Pôle national des données de biodiversité” (PNDB) et financé par le “réseau des acteurs du Développement LOGiciel au sein de l’Enseignement Supérieur et de la Recherche” (DevLOG).
Vidéos en anglais sur le sujet : « Transforming scattered analyses into a documented, reproducible and shareable workflow – FOSDEM Brussels », 2020-02 et The “Rmd first” method: when projects start with documentation – useR Toulouse, 2019-07
Sommaire
Le projet “Vigie-chiro”
Lors de cet événement, j’ai présenté les bonnes pratiques de développement R, avec la méthode “Rmd first” (cf. Rmd first : Quand le developpement commence par la documentation). Nous avons ensuite discuté de mettre en forme les scripts du projet de science participative “Vigie-Chiro”.
Le projet “Vigie-Chiro” s’inscrit dans le programme Vigie-Nature. Il s’agit d’un projet de suivi des populations de chauves-souris. Chacun peut participer à ce projet de sciences participatives (Plus d’information sur le site web).
Cet article de blog présente la démarche que nous avons adoptée pour transformer des scripts R éparpillés en un beau package.
Accepter que son travail soit décortiqué
Ce format “hackathon” a obligé un chercheur à présenter son code à tout le monde.
Je me sens nu d’avoir présenté mes scripts.
Ce n’est pas facile de se “mettre à nu” en présentant ses scripts à d’autres personnes, en particulier des inconnus lors d’un hackathon, et surtout quand ils sont dans la même pièce. Les développeurs présents lors de ces “fêtes collaboratives” sont (normalement) bienveillants, donc il y a des chances que ça se passe bien. Si on a partagé un repas ensemble la veille, c’est aussi un peu moins impressionnant. Mais ça fait un gros changement par rapport à travailler seul, et ce n’est pas du tout la même chose que de présenter les résultats de ses analyses pour une réunion, une conférence ou une publication.
État des lieux du projet avant le hackathon
Le projet est déjà versionné sur Github, ce qui est un très bon début. Il contient de nombreux scripts R, certains ont des fonctions, d’autres sont des scripts bruts. Les scripts principaux avec les fonctions, ont récemment été déplacés dans un dossier “functions”. Tous les autres scripts sont à la racine du projet, et ils ne sont pas forcément nécessaires au flux de travail ciblé.
L’état du projet git sur Github au démarrage :
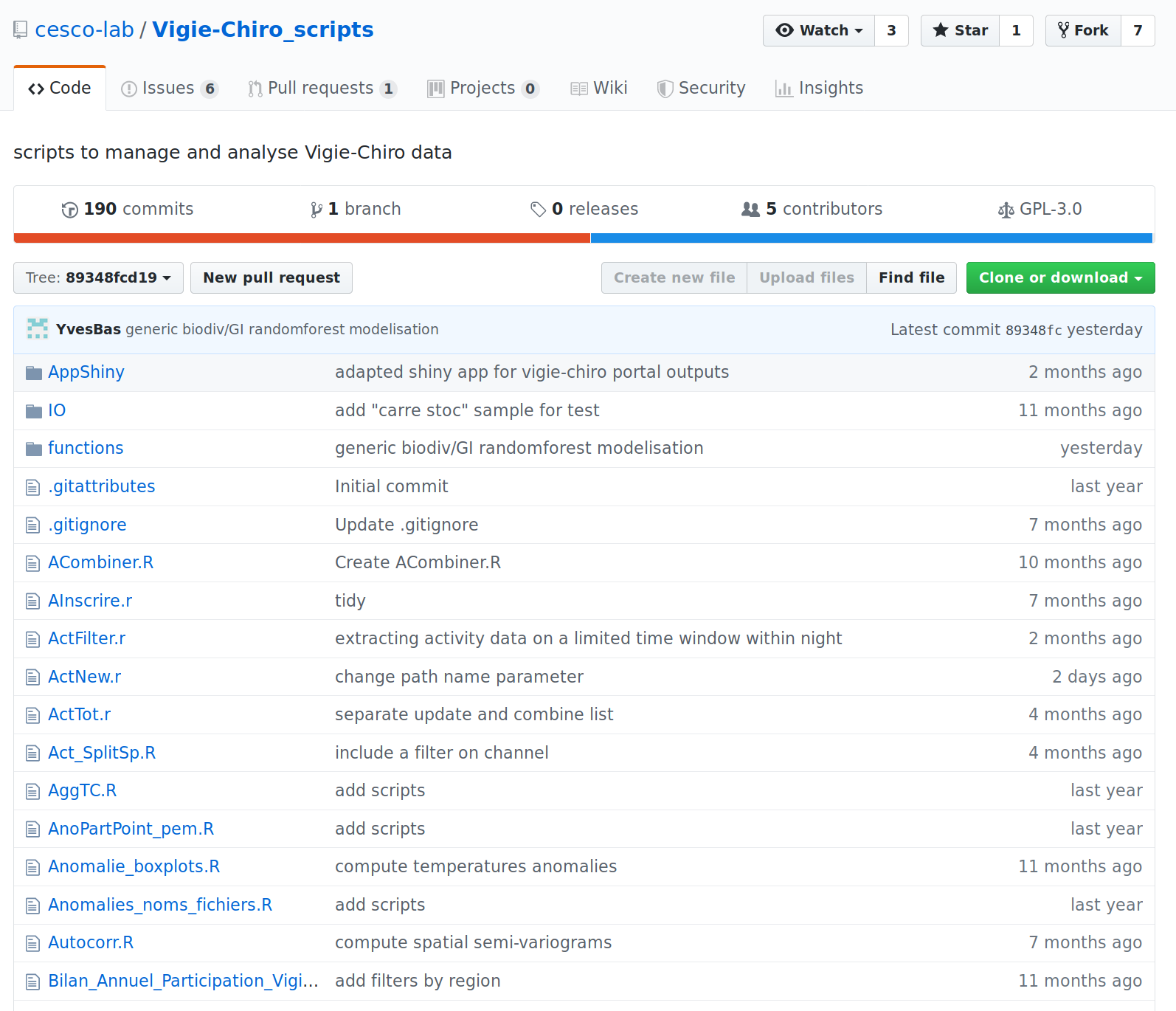
Déblayer l’existant et clarifier l’articulation des scripts
Je pensais que ça n’allait jamais commencer, que ça allait être très compliqué avec tout ce qu’il y avait à faire. Et finalement, les échanges sont très intéressants, on s’est mis à coder, et on a avancé pas mal de choses.
Ça prend du temps de faire le tri dans un dossier de 30 scripts, d’expliquer à des personnes qui ne connaissent ni les données, ni les objectifs la façon dont ils sont actuellement utilisés, et vers où on veut aller. Quand on a une bonne vision de la façon de gérer ce genre de projet, c’est plus efficace. L’objet de cet article de blog est de vous aider à diriger ces discussions et à structurer un travail collaboratif.
Les scripts du projet “Vigie-Chiro” que nous conservont permettent de prédire la distribution d’espèces de chauves-souris en fonction de variables environnementales.
Mettre à plat le flux de travail
Prenons un tableau et dessinons les liens entre les scripts.
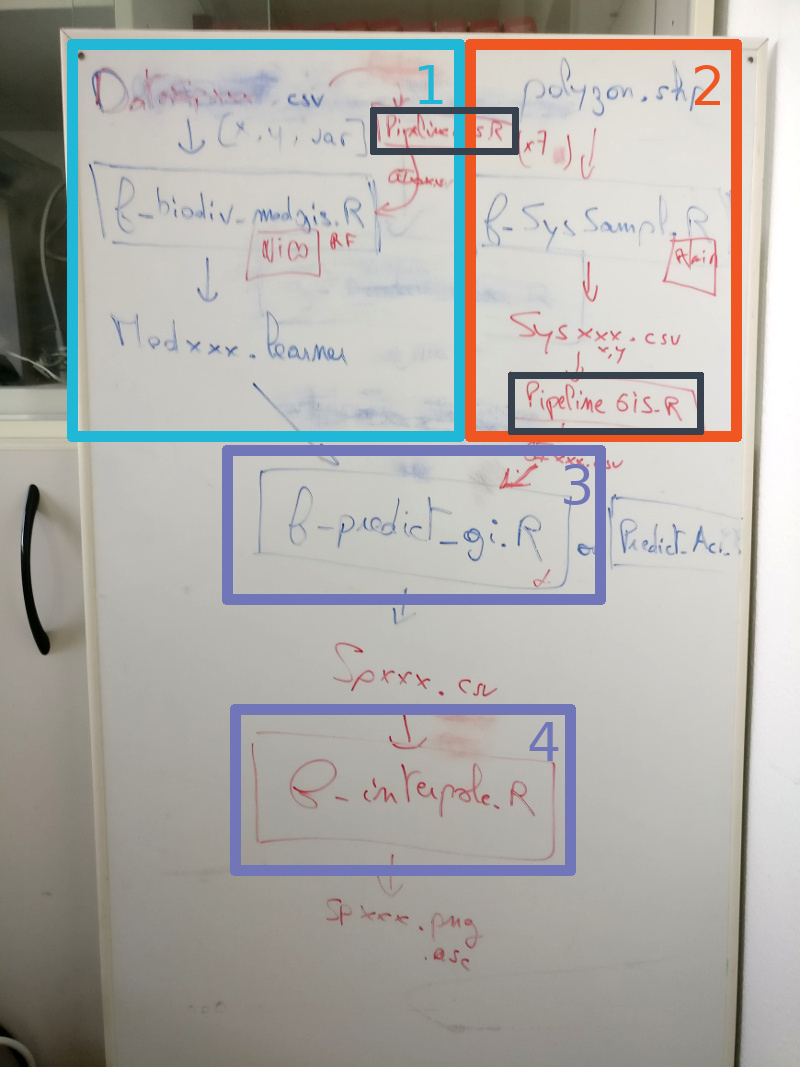
Le flux qui va de la donnée jusqu’à la prédiction commence dans 2 parties distinctes qui se rejoignent pour la prédiction. Une étape préalable au projet est la récupération de rasters de variables environnementales en tout genre couvrant la France Métropolitaine.
- Les stations d’observation
- Nous faisons l’extraction des valeurs des variables environnementales pour les stations d’observation
- Un modèle, de type forêt aléatoire ici, est ajusté sur les données d’observations pour estimer la probabilité de présence en fonction des variables environnementales
- Les stations de prédiction
- Nous construisons une grille regulière sur une partie de la zone d’étude qui servira de grille de prédiction
- L’extraction des variables environnementales est effectuée au niveau de ces points de prédiction
- La prédiction
- Le modèle ajusté est utilisé pour prédire les probabilités de présence au niveau des points de prédiction
- La création des cartes de prédiction
- Dessiner les cartes
L’étape d’extraction des variables environnementales se fait avec un script transverse, commun aux deux branches: “Pipeline_GIS.R”.
Retour d’expérience
Lorsqu’on n’en a jamais fait, un package peut faire peur. Pour réduire ses craintes, il faut se dire que ce n’est ni plus ni moins qu’un dossier organisé d’une manière contrainte. Cette organisation permet a tout développeur·se de package de s’intégrer à un projet, car il·elle est dans une configuration connue.
Toutefois, ici, nous sommes passés de l’utilisation de multiples scripts rangés dans différents dossiers à la mise en package. Certain·e·s n’avaient jamais fait de package. Nous avons ajouté par-dessus, la découverte de la gestion d’un projet git à plusieurs développeurs·euses. Pour réduire la hauteur de la marche, nous avons commencé à développer à partir de scripts R, dans un projet qui n’est pas un package.
Ne pas passer directement en package a rapidement montré quelques inconvénients :
- La lecture de données se fait avec l’utilisation de la fonction
file.path(), voire avec l’utilisation du package {here} si les données ne sont pas stockées dans un dossier à l’intérieur du dossier de vignettes. Ce qui est fortement recommandé.- De fait, pour mimer le futur package, les données peuvent être stockées dans un dossier “inst/example-data”, qui sera repéré avec
here::here("inst/example-data/my_data.csv").
- De fait, pour mimer le futur package, les données peuvent être stockées dans un dossier “inst/example-data”, qui sera repéré avec
- Les fonctions créées ne peuvent être appelées qu’avec l’utilisation de la fonction
source()dans les scripts ou les Rmarkdown, avec des chemins nécessitant aussi {here}.- Ces appels à
source()ne sont pas nécessaires dans le cadre d’un package puisque les fonctions sont référencées lors du chargement du package.
- Ces appels à
Afin d’éviter ces inconvénients et éviter d’avoir à repasser sur tous les scripts pour s’adapter au package, vous devriez directement transformer votre dossier en package. N’en ayez pas peur ! Cela vous permet aussi de vérifier régulièrement que le package passe bien les tests de conformité.
Les étapes de transformation vers le package
Ré-organiser le dossier
Un package a une structure bien définie. Les scripts R qui contiennent les fonctions du package doivent être rangées dans un dossier nommé “R”. Nous avons par ailleurs convenu que le meilleur moyen de diffuser au mieux ce package serait de créer une ou plusieurs vignettes en guise de guide d’utilisation des fonctions et du flux de travail. C’est une méthode “Rmd-first” adaptée à la présence de fonctions déjà existantes. Dans un package, les vignettes sont stockées dans un dossier nommé “vignettes”.
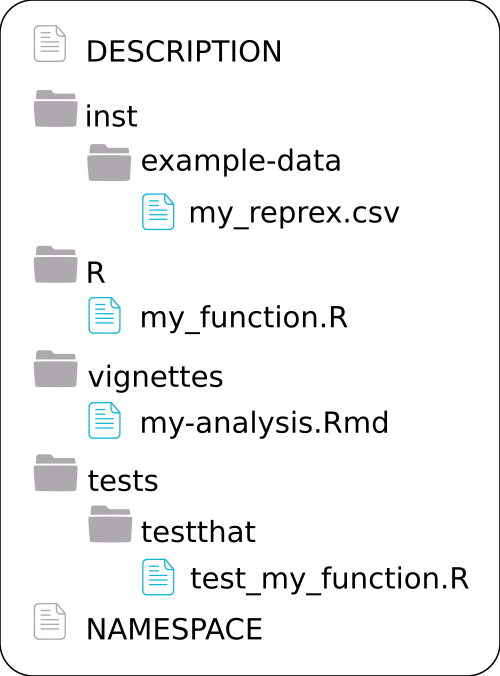
La stratégie recommandée est :
- Stocker tous les scripts R et autres fichiers dans un dossier “misc”, caché du futur package par un “.Rbuildignore”:
usethis::use_build_ignore("misc") - Transformer en package R
- Transformer les scripts un par un en fonctions documentées
- Tester les fonctions avec des exemples reproductibles
- Rédiger les vignettes d’explication de l’utilisation des fonctions avec les exemples reproductibles, en parallèle des exemples testés
- Déplacer les fonctions dans le dossier R au fur et à mesure
Transformer en package R
Vous avez lu les inconvénients dus à une mise en package trop tardive. Vous devriez donc commencer directement par la transformation en package. Quelque soit l’ordre des opérations, vous serez de toutes façons obligé·e·s de passer par ces étapes :
- Changer le nom du dossier du projet s’il n’est pas conforme aux règles de noms de package (pas de caractères spéciaux, seuls les
.sont autorisés), e.g. “mon.package” - Utiliser
usethis::create_package("mon.package")en dehors de votre répertoire de travail - Remplir le fichier DESCRIPTION
- Gérer la licence avec
usethis::use_xxx_license("Mon Nom") - Cacher tous les fichiers inutiles (pour le moment) au package, comme avec
usethis::use_build_ignore("misc") - Stocker ces commandes de formattage du package dans un script R à la racine, nommé
dev_history.R, aussi caché avecusethis::use_build_ignore("dev_history.R") - Le dossier “R” étant vide, on peut déjà vérifier le package avec
devtools::check().- Vous aurez sûrement quelques erreurs, warnings et autres notes à gérer, mais un petit tour sur Internet vous aidera à comprendre les messages compliqués
- Prenez aussi l’habitude de faire passer la fonction
attachment::att_amend_desc()(par défaut, sans paramètre) pour gérer la documentation et les dépendances
Je vous renvoie, de nouveau, vers l’article “Rmd first : Quand le developpement commence par la documentation” pour plus détails.
Transformer les scripts en fonctions
- Il convient d’identifier le grand rôle de chacun des scripts. Chacun de ces rôles finira en une grosse fonction R. Dans notre cas, nous avons les quatre étapes identifiées précédemment ainsi que le script transverse, donc cinq scripts.
- Pour chacun de ces scripts, il faut faire remonter tout ce qui est ou peut être un paramètre modifiable par l’utilisateur·trice ou dépendant de l’utilisateur·trice. En particulier les chemins d’accès aux données doivent être des paramètres de la future fonction.
- Aucun chemin ne doit être spécifié de manière absolue dans une fonction, sauf si c’est un chemin vers un jeu de données interne au futur package qui peut être appelé avec
system.file().
- Aucun chemin ne doit être spécifié de manière absolue dans une fonction, sauf si c’est un chemin vers un jeu de données interne au futur package qui peut être appelé avec
- La documentation des fonctions se fait au fur et à mesure en ajoutant le squelette {roxygen2} définissant le titre, les paramètres nécessaires (
#' @param) et les dépendances (#' @importFrom)
Pour commencer, il n’est pas nécessaire de découper les scripts en de multiples fonctions R. C’est quelque chose qui peut se faire dans un second temps pour faciliter la maintenance.
Déplacer les fonctions dans le dossier R
Une fois créées et documentées, les fonctions peuvent passer dans le dossier R afin d’être testées et vérifiées.
- Générer la documentation avec
attachment::att_amend_desc() - Charger les fonctions dans l’environnement de travail avec
devtools::load_all()
Créer des exemples reproductibles
La mise en fonction des scripts nécessite de pouvoir vérifier que nous avons travaillé correctement en testant des données d’exemple. Nous devons pouvoir vérifier que les modifications apportées et la mise en paramètre n’affecte pas le résultat. Pour cela, il est fortement recommandé de travailler avec des petits exemples reproductibles. Ils peuvent être totalement fictifs ou être une extraction des données utilisées habituellement sur ces scripts. On peut même combiner les deux approches. Plus il y a d’exemples testés, plus on peut être confiant sur nos scripts.
- La création de ces exemples reproductibles permet de compléter la documentation en ajoutant le tag
#' @examplesdans le squelette {roxygen2} des fonctions - L’exemple reproductible peut (doit) aussi servir à la création de tests unitaires avec {testthat}. C’est extrêmement utile pour la maintenance du package, mais aussi pour un développement tel que celui-ci avec plusieurs développeurs·ses en parallèle qui peuvent modifier, sans s’en rendre compte, les comportements des fonctions dépendantes les unes des autres.
- Les données internes (stockées dans “inst/example-data” par exemple) peuvent être retrouvées dans les exemples et tests avec
system.file("example-data/mes_data.csv", package = "my.package")

Dans notre cas, nous avons des données intermédiaires qui permettent de tester la validité de chacun des scripts, quelque soit l’ordre dans lequel on décide de réaliser la mise en fonctions. C’est un point très intéressant pour le “collaboration fest” car nous pouvons répartir le travail entre différents développeurs, chacun sur sa fonction, sans dépendance ou potentiel conflit entre les fichiers scripts créés. Ces données sont stockées dans “inst” mais son trop lourdes pour être raisonnablement laissées dans le package, voire même trop lourdes pour être envoyées sur GitHub/Gitlab. Elles servent au développement pour vérification, mais doivent être remplacées par des exemples reproductibles plus restreints.
Créer des vignettes / guide d’utilisation
La création de la vignette peut se faire dès le développement et la mise en place des tests de notre fonction. Un texte doit présenter les données utilisées pour l’exemple, expliquer l’objectif de chacune de fonctions, puis la façon de les utiliser. On y présente les valeurs des paramètres avec les variations des résultats qu’ils entraînent.
Dans notre cas, afin notamment d’éviter les conflits de fichiers, nous sommes partis sur une vignette pour chacune des parties du flux d’analyse. À terme, une vignette unique pourra présenter le flux complet de l’analyse. Elle pourra ainsi être utilisée comme script de référence unique pour que les utilisateurs·trices puissent réaliser leurs analyses sur leurs propres données. Les vignettes intermédiaires seront toutefois conservées, notamment pour de la documentation plutôt orientée pour les développeurs·euses.
Finalisation
- A chaque étape, il convient de regénérer la documentation, faire passer les tests pour éviter d’avoir à chercher la source des problèmes après de trop nombreuses modifications.
- Partagez, testez avec de nouveaux jeux de données.
- Au cours de l’évolution de l’utilisation, vous pouvez prendre du temps pour subdiviser les grosses fonctions en unités plus petites.

J’ai créé un exemple reproductible minimal avec un seul point, ce cas particulier fait planter la fonction à plusieurs endroits du script, non pas parce que l’exemple est mal choisi, mais parce que la fonction n’a jamais été testée pour ce cas extrême. Pourtant, ce pourrait être un cas possible.
Un package est en développement permanent. Vous vous rendrez compte de cas particuliers qui nécessitent des modifications des fonctions. Lors de ces différents problèmes, n’hésitez pas à ajouter des tests à l’intérieur des fonctions (e.g. if(test is TRUE) {stop("message explicite")}). Et comme vous vérifiez les changements sur un petit reprex, vous pouvez ajouter de nouveaux tests unitaires en parallèle.
Un point sur la gestion de projet avec git
git est un outil indispensable pour la gestion d’un projet de développement, seul ou à plusieurs. Il permet le versionnement des fichiers et le développement en parallèle de plusieurs fonctionnalités. Il gère la fusion des fonctionnalités dans la branche principale (master). Par ailleurs, avec une interface comme Gitlab ou GitHub, il est possible de faire de la gestion de projet.
Dans notre cas,
- Nous avons créé des issues sur Github pour matérialiser les différentes fonctionnalités à développer. Ici, l’une des cinq fonctions du package, ainsi que des tâches de mise en package.
- Chaque développeur·se crée un fork du projet principal sur Github.
- Les développements de chacun·e se font en local dans des branches de leur fork.
- On ne modifie jamais directement le master.
- La branche master de son fork doit rester à jour du master du dépôt original (cf mon article sur la gestion des master entre fork).
- Lorsqu’une fonctionnalité est terminée, le·la développeur·se crée un commit mentionnant le numéro de l’issue.
- Il peut proposer de fermer l’issue lors de l’intégration dans le master: e.g.
closes #4.
- Il peut proposer de fermer l’issue lors de l’intégration dans le master: e.g.
- Il peut alors proposer une pull/merge request vers le dépôt principal, que le·la mainteneur peut accepter en l’état ou pour laquelle il·elle peut demander des modifications.
Publiez votre code
Mon passé de chercheur resurgit…
Si vous êtes dans le milieu de la recherche, vous courez après les publications et les citations de votre travail. Pensez-bien que la citation ne s’arrête pas aux questions biologiques ou écologiques. Un jeu de données est un matériel publiable et citable. Un code est un matériel publiable et citable. Ne réduisez plus votre partie analyse de données, créez des codes, ou des packages R comme ici, qui soient documentés et ré-utilisables par d’autres scientifiques. Et pour être cités, il faut penser à leur attribuer un DOI. Pour celà, rien de plus simple si vous avez déjà mis votre code sur Github, utilisez la plateforme européenne Zenodo. Vous vous connectez avec votre compte Github et Zenodo attribue un DOI aux dépôts Github que vous avez choisis, dès lors que vous créez un release. A chaque nouvelle version (release), vous aurez un nouveau DOI, avec archivage de votre code en l’état et une citation associée. Pour un package R, vous pouvez ajouter cette citation dans un fichier “inst/CITATION”.
Pour exemple, je l’ai fait avec mon package {SDMSelect}: Cross-validation model selection and species distribution mapping, disponible sur Github uniquement. Il y a un badge Zenodo cliquable sur le README et le fichier CITATION est le suivant :
citHeader("To cite SDMSelect in publications use:")
citEntry(entry = "misc",
title = "SDMSelect: A R-package for cross-validation model selection and species distribution mapping.",
journal = "Zenodo",
year = "2017",
url = "http://doi.org/10.5281/zenodo.894344",
textVersion =
paste("Sébastien Rochette. (2017, September 18).",
"SDMSelect: A R-package for cross-validation model selection and species distribution mapping.",
"Zenodo.",
"http://doi.org/10.5281/zenodo.894344")
)Vous trouverez plus d’informations (en anglais) sur la façon d’utiliser Github avec Zenodo sur Making Your Code Citable

Station marine de Concarneau – Muséum National d’Histoire Naturelle
A vous de jouer !
Les packages cités dans cet article: {usethis}, {attachment}, {golem}, {here}, {devtools}, {SDMSelect}.
Pour un accompagnement sur la mise en package de vos projets ou des formations aux bonnes pratiques de développement, seul ou à plusieurs, n’hésitez pas à faire appel à nous.





Laisser un commentaire