Nous revoilà pour le troisième volet de notre série SAS2R, avec un sujet de la plus haute importance : la manipulation de données. Si vous êtes arrivés ici, c’est que vous avez sûrement l’habitude de jongler avec tout un tas de tables sas, d’en faire des sous-sélections, des merges, et autres réjouissances, que ce soit avec des étapes DATA, ou encore des PROC SQL. Et ce qui est top, c’est qu’après avoir lu cet article, vous saurez faire tout ça dans R aussi !
Note : Dans cet article, je vous présenterai deux manières distinctes de procéder en R. La première est d’utiliser les fonctions de base de R, qui ne nécessitent aucun chargement de package particulier. La seconde, et celle que je vous recommande fortement, est l’utilisation des fonctions du {tidyverse} qui présente de nombreux avantages comme la lisibilité de sa syntaxe ou l’homogénéité des différents packages du {tidyverse}.
Sommaire
SAS vs. R – lignes vs. colonnes
Une petite digression avant d’entrer dans le vif du sujet, mais il me semblait intéressant de commencer par noter ce qui distingue SAS et R, dans la manière même de raisonner. En effet, ces deux langages adoptent des logiques complètement différentes. Je m’explique : l’étape DATA de SAS fonctionne ligne par ligne, quand R est plutôt orienté colonne/vecteur. Un exemple simple :
Soit la table tab_input suivante :
| v1 | v2 | v3 |
|---|---|---|
| 1 | 4 | 7 |
| 2 | 5 | 8 |
| 3 | 6 | 9 |
Je souhaite par exemple calculer la somme cumulée de v2.
Considérons maintenant l’étape DATA suivante, qui permet de faire cela :
DATA tab_output ;
RETAIN Cumsum 0 ;
SET tab_input ;
Cumsum=cumsum+v2 ;
RUN ;Note : ici RETAIN permet de retenir la valeur de total à chaque ligne.
En regardant pas à pas ce que fait SAS, cela donne :
- création de la variable cumsum qui vaut 0 dans le vecteur de travail

- la première ligne de tab_input est recopiée

- la valeur de cumsum est incrémentée de la valeur de v2

RUNenregistre le vecteur de travail dans la première ligne de la table tab_output et réinitialise le vecteur de travail

- itération sur toutes les autres lignes de la table tab_input, pour finalement obtenir la table tab_output suivante :
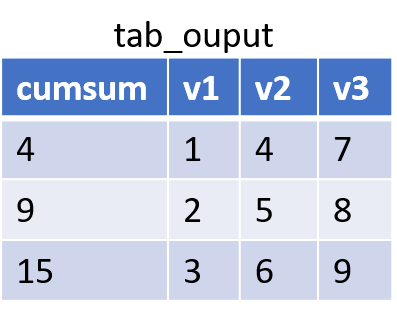
Je pense qu’on est tous d’accord sur un point : c’est fastidieux ! Et dans R, ça se passe comment du coup ? Considérons le code suivant :
# version R-base
tab_output <- tab_input
tab_output$cumsum <- cumsum(tab_ouput$v2)
# version {tidyverse}
library(dplyr)
tab_output <- tab_input %>% mutate(cumsum = cumsum(v2))La somme cumulée de v2 est calculée en une seule fois puis ajoutée à la table tab_output dans la variable cumsum, ajoutée après les autres variables. Notez que nous aurions pu créer la nouvelle colonne directement dans la table originale, mais il est fortement recommandé de ne jamais modifier des données originales. Cela vous permet aussi de choisir un nouveau nom, plus approprié au contenu de cette nouvelle table plus complète. La table obtenue sera la suivante :
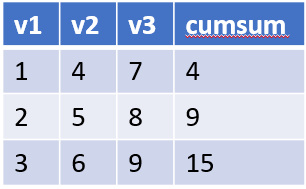
Note : Attention, contrairement à SAS, R est sensible à la casse. Nous avons nommé la nouvelle colonne cumsum, en minuscules, pour respecter les bonnes pratiques de code en R. D’une manière générale, comme R est sensible à la casse, il vaut mieux tout écrire en minuscules comme ça vous n’avez pas à tenter de vous souvenir où vous avez mis des majuscules… Notez que cumsum() est une fonction qui calcule la somme cumulée d’un vecteur donné.
Tour d’horizon : ce que DATA a à offrir, ce qu’apporte la PROC SQL
L’étape DATA est primordiale en SAS. Elle permet en effet :
- l’import et la création de données
- la sélection de lignes et de colonnes qui nous intéressent
- la modification des attributs d’une variable/colonne
- la concaténation et la fusion de tables
- de faire des boucles et des tests
- d’appliquer des fonctions
- la création de nouvelles variables
- …
Et la PROC SQL dans tout ça ? Grosso-modo, elle permet de faire à peut près les mêmes choses au niveau de la sous-sélection de données et de la fusion de tables, mais cette fois à l’aide du langage de requête de bases de données relationnelles, et avec quelques avantages :
- la fusion de deux tables ne requiert pas que les noms de variables soient identiques
- le tri préalable des données via une PROC SORT n’est pas nécessaire
- plus simple et plus rapide d’utilisation
Import et création de jeux de données
La recopie d’une table
Pour créer une nouvelle table à partir d’une table déjà existante, dans SAS, avec une étape DATA :
DATA data_destination;
SET data_source;
RUN;En passant par une PROC SQL :
PROC SQL;
SELECT * as data_output
FROM data_input
QUIT;Dans R :
data_destination <- data_sourceNotez que R crée une copie de la table originale. Si vous modifiez le jeu data_source a posteriori, il n’y aura aucune répercution sur data_destination, à moins de relancer cette ligne de code.
La saisie de données
Dans SAS, il est possible de créer une table en saisissant des données, via les instructions INPUT qui permet de déclarer les noms des variables, et CARDS qui permet d’entrer les données. L’instruction FORMAT permet de spécifier le format des variables. L’utilisation de $ indique un format alphanumérique. Quand le format n’est pas spécifié, la variable est numérique.
DATA table1;
CARDS;
tutu M 170 1982-12-11
toto M 182 1982-12-21
titi F 157 1982-12-25
RUN;Pour créer cette table en R-base :
table1 <- data.frame(
nom = c("tutu", "toto", "titi"),
sexe = c("M", "M", "F"),
taille = c(170, 182, 157),
format = "%Y-%m-%d")
)
# Afficher la table
table1
## 1 tutu M 170 1982-12-11
## 2 toto M 182 1982-12-21
## 3 titi F 157 1982-12-25Une fois de plus, on note cette logique opposée : les tables SAS se construisent ligne par ligne, alors qu’en R on construit une table colonne par colonne.
Cependant, en plus de proposer une fonction qui contruit une table par colonnes (tibble(), plus résilient que data.frame()), le package {tibble} du {tidyverse}, dispose également d’une fonction permettant de construire une table par lignes (tribble()) :
library(tibble)
# construction par colonnes :
table1 <- tibble(
nom = c("tutu", "toto", "titi"),
sexe = c("M", "M", "F"),
taille = c(170, 182, 157),
format = "%Y-%m-%d")
)
# construction par lignes :
table1 <- tribble(
"tutu", "M", 170, as.Date("1982-12-11", format = "%Y-%m-%d"),
"toto", "M", 182, as.Date("1982-12-21", format = "%Y-%m-%d"),
"titi", "F", 157, as.Date("1982-12-25", format = "%Y-%m-%d")
)L’import de données
En SAS, l’import des données externe provenant d’un fichier texte (“.txt” avec colonnes séparées par des espaces) se fait grâce à l’instruction INFILE. Le paramètre dlm permet de renseigner le séparateur, et firstobs le numéro de ligne de la première observation; si les noms des variables constituent la première ligne de nos données, on choisira firstobs=2. L’instruction INPUT permet de spécifier les noms de variables, et cela peut être laborieux, imaginez que vous ayez des dizaines de colonnes !
(L’import de données autres que textes se fait grâce à la PROC IMPORT, mais ce n’est pas le sujet d’aujourd’hui)
DATA table2;
INFILE ’/chemin/donnees.txt’ dlm=’ ’ firstobs=1;
INPUT nom $ sexe $ taille ;
RUN;La fonction la plus utilisée dans R pour l’import de données est la fonction read.table(). Si on souhaite importer les données :
table2 <- read.table(file = "chemin/donnees.txt", header = FALSE, sep = " ")
colnames(table2) <- c("nom", "sexe", "taille")Mais si le nom des variables est présent dans les données, il suffit d’écrire :
table2 <- read.table(file = "chemin/donnees.txt", header = TRUE, sep = " ")Regardons de plus près les paramètres les plus utiles de la fonction read.table() :
file: le chemin du fichierheader:TRUEsi les données contiennent les noms de variables,FALSEsinonsep: le séparateurdec: le caractère de décimalerow.names:TRUEsi les données contiennent les noms des lignes,FALSEsinonna.strings: la chaîne de caractères correspondant aux valeurs manquantes, à interpréter commeNAstringsAsFactors:TRUEsi les variables alphanumériques doivent être considérées comme catégorielles (ou facteur),FALSEsinon
Dans notre exemple, la variable nom n’est pas un facteur, mais la variable sexe si. On peut donc écrire :
table2 <- read.table(file = "chemin/donnees.txt", header = TRUE, sep = " ", stringsAsFactors = FALSE)
table2$sexe <- as.factor(tables2$sexe)Pour les autres paramètres, consultez l’aide de la fonction comme suit :
?read.tableDe la même manière, vous pouvez utiliser les fonctions read.delim() et read.delim2() avec les mêmes arguments.
Puisqu’ici le but est de traduire l’étape DATA en R, je ne m’attarderai pas sur l’import des données autres que les fichiers textes, mais je vous incite fortement à aller regarder les fonctions read.csv(), read.csv2(), ou encore read.xlsx() du package {xlsx}. Pour les jeux de données plus lourds, la fonction fread() du package {data.table} peut être intéressante.
Si vous avez fait le (bon) choix de préférer le {tidyverse} au R-base, bonne nouvelle, le package {readr} dispose des fonctions dont nous avons besoin pour importer tous types de données : read_csv(), read_tsv(), read_delim(), read_fwf(), read_table(), read_log(). En reprenant l’exemple précédent :
library(dplyr) # pour la fonction %>%, qui peut être traduite par 'et ensuite', et mutate() ici
library(readr)
table2 <- read_table(file = "chemin/donnees.txt", col_names = TRUE) %>%
mutate(sexe = as.factor(sexe))Regardons de plus près les paramètres les plus utiles de la fonction read_table() :
file: le chemin du fichiercol_names:TRUEsi les données contiennent les noms de variables,FALSEsinonna: la chaîne de caractères correspondant aux valeurs manquantes, à interpréter commeNA
Attention, cette fonction ne permet pas de spécifier le séparateur, dans le fichier les colonnes sont séparées par un ou plusieurs espaces. Si vous cherchez l’équivalent en spécifiant le délimiteur, regardez read_delim(). Choisissez bien votre fonction d’import de données en fonction de leur nature.
Je vous rappelle par ailleurs que nous avons déjà évoqué dans le premier article de cette série l’import de données SAS au format sas7bdat ou xpt .
Sélection de lignes et de colonnes
La sélection de colonnes
L’étape DATA permet de ne sélectionner qu’une partie des colonnes d’une table donnée, grâce aux instructions DROP et KEEP. Prenons l’exemple d’une table à 5 variables, pour laquelle on ne souhaite garder que les 3 premières. Ces 4 façons d’écrire donnent le même résultat :
/* Avec l'instruction KEEP */
DATA data_output;
SET data_input;
KEEP v1 V2 v3;
RUN;
DATA data_output;
SET data_input (KEEP=v1 V2 v3);
RUN;
/* Avec l'instruction DROP */
DATA data_output;
SET data_input;
DROP v4 v5;
RUN;
DATA data_output;
SET data_input (DROP=v4 v5);
RUN;Avec la PROC SQL, on a :
PROC SQL;
SELECT v1, v2, v3 as data_output
FROM data_input;
QUIT;En R, la sélection de colonnes peut se faire de différentes manières :
# par le nom des variables :
data_output <- data_input[,c("v1", "v2", "v3")]
# ou encore :
data_output <- data_input[,-c("v4", "v5")]
# par les numéros de colonnes :
data_output <- data_input[,c(1, 2, 3)]
# ou encore :
data_output <- data_input[,-c(4, 5)]La fonction select() du package {dplyr} permet également d’opérer une sélection sur les colonnes :
library(dplyr)
# par le nom des variables :
data_output <- data_input %>% select(v1, v2, v3)
# ou encore
data_output <- data_input %>% select(-v3, -v4)
# par les numéros de colonnes :
data_output <- data_input %>% select(1, 2, 3)
# ou encore
data_output <- data_input %>% select(-4, -5)
# ou un mix des deux
data_output <- data_input %>% select(v1, 2, v3)Notez qu’il n’est pas recommandé de sélectionner sur la position. En effet, il y a plus de risques de déplacer les variables dans un jeu de données initial que de les renommer. Aussi, avec les positions, votre analyse ne serait plus reproductible. Préférez la sélection par le nom.
De fait, la fonction select() permet d’aller plus loin encore dans la sélection des colonnes. Prenons par exemple les données iris :
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosaJe souhaite obtenir les tables suivantes :
tab1qui ne contient que les variables dont le nom commence par'Sepal'tab2qui ne contient que les variables dont le nom termine par'Length'
Les fonctions starts_with() (“commence par”) et ends_with() (“se termine par”) appelées en paramètres à la fonction select() nous permettent cela :
tab1 <- iris %>% select(starts_with("Sepal"))
head(tab1)## Sepal.Length Sepal.Width
## 1 5.1 3.5
## 2 4.9 3.0
## 3 4.7 3.2
## 4 4.6 3.1
## 5 5.0 3.6
## 6 5.4 3.9tab2 <- iris %>% select(ends_with("Length"))
head(tab2)## Sepal.Length Petal.Length
## 1 5.1 1.4
## 2 4.9 1.4
## 3 4.7 1.3
## 4 4.6 1.5
## 5 5.0 1.4
## 6 5.4 1.7La sélection de lignes
Si on se trouve dans le cas où l’on connait le numéro des lignes que l’on souhaite garder, et que ces lignes sont consécutives, on utilise les options FIRSTOBS et OBS de l’instruction SET. On souhaite par exemple garder les lignes comprises entre 10 et 20 incluses :
DATA data_output;
SET data_input (FIRSTOBS=10 OBS=20);
RUN;Dans R, cela se traduit de la façon suivante :
# R-base :
data_output <- data_input[10:20,]
# tidyverse :
library(dplyr)
data_output <- data_input %>% slice(1:20)Mais en général, on ne sait pas quels sont les numéros des lignes à garder, surtout si on a au préalable trié les données. D’ailleurs, en théorie, on ne filtre pas des individus sans raison, normalement, le choix des lignes ou observations à conserver se fait conditionnellement aux valeurs d’une ou plusieurs variables. Sinon, c’est qu’il doit manquer une variable dans vos données…
Dans SAS, cela est possible via l’instruction WHERE de l’étape DATA. Par exemple, si on a un jeu de données data_input contenant au moins les colonnes v1 et v2 et que l’on souhaite sélectionner les lignes pour lesquelles v1 a une valeur positive et v2 a pour valeur “a” :
DATA data_output;
SET data_input;
WHERE v1>0 AND v2="a";
RUN;Et en passant par une PROC SQL :
PROC SQL;
SELECT * as data_output
FROM data_input
WHERE v1>0 AND v2="a";
QUIT;En R, cela donne :
data_output <- data_input[data_input$v1 > 0 & data_input$v2 == "a",]Encore une fois, le package {dplyr} nous propose plusieurs fonctions utiles permettant de faire des filtres sur les observations :
- la fonction
filter(). Si on reprend notre dernier exemple, cela donne :
data_output <- data_input %>% filter(v1 > 0 & v2 == "a")
# ou bien :
data_output <- data_input %>% filter(v1 > 0, v2 == "a")- la fonction
filter_all()permet d’aller plus loin en conditionnant la sélection sur toutes les variables. Supposons une tabletab_xdexvariables numériques.
# sélection des lignes pour lesquelles toutes les variables ont une valeur supérieure à 50 :
tab_x %>% filter_all(all_vars(. > 50)) # -> all_var() correspond au AND, l'intersection.
# sélection des lignes pour lesquelles au moins une variable a une valeur supérieure à 50 :
tab_xtab_x %>% filter_all(any_vars(. > 50)) # -> any_vars() correspond au OU, l'union.- la fonction
filter_at()permet quant à elle la même chose, mais sur une sélection de variables :
# sélection des lignes pour lesquelles toutes les variablescommençant par "d" ont une valeur supérieure à 50 :
tab_x %>% filter_at(vars(starts_with("d")), all_vars(. > 50)) - Avec la nouvelle version de {dplyr} (>= v1.0.0), nous utiliserons
accross(). (Voir Hey ! Quoi de neuf {dplyr} ? Le point sur la v1 !)
# sélection des lignes pour lesquelles toutes les variablescommençant par "d" ont une valeur supérieure à 50 :
tab_x %>% filter(accross(starts_with("d"), all_vars(. > 50))) Modification des attributs des variables
Dans SAS, les variables ont certains attributs obligatoires :
- le nom
- la longueur de stockage
- le type
Et d’autres optionnels :
- le label (qui s’il est spécifié sera affiché à la place du nom de la variable)
- le format d’écriture (ide mque le format de lecture, mais qui sert lors de l’écriture de la table)
Si pour connaître les attributs des variables d’une table en SAS vous utilisez la PROC CONTENTS, dans R plusieurs solutions s’offrent à vous. Reprenons le jeu de données iris vu précédemment :
# pour connaitre les noms des variables :
names(iris)## [1] "Sepal.Length" "Sepal.Width" "Petal.Length" "Petal.Width" "Species"# pour connaitre les types des variables :
str(iris)## 'data.frame': 150 obs. of 5 variables:
## $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
## $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
## $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
## $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...Pour modifier l’attribut nom d’une variable dans SAS dans une étape DATA, on utilise l’instruction RENAME :
DATA data_output;
SET data_input;
RENAME ancien_nom=nouveau_nom;
RUN;Dans R, on peut procéder comme suit :
data_output <- data_input
names(data_output)[which(names(data_output) == "ancien_nom")] <- "nouveau_nom"
# which(names(data_output) == "ancien_nom") détermine l'indice de la colonne à modifier.
# si on sait que la variable à modifier est la 3ème de la table, on écrira alors :
names(data_output)[3] <- "nouveau_nom"En utilisant le {tidyverse}, si l’on souhaite renommer une variable, on utilise la fonction rename() du package {dplyr} :
library(dplyr)
data_output <- data_input %>% rename(nouveau_nom = ancien_nom)En ce qui concerne les modifications de formats de variables dans R, voici quelques unes des fonctions les plus utilisées :
as.factor()pour transformer une variable chaine de caractères en variable facteuras.numeric()pour transformer une variable en variable numérique
Génération de variables
L’étape DATA de SAS permet de créer de nouvelles variables et de les ajouter à une table donnée. De par son fonctionnement, SAS ajoute les données d’une nouvelle colonne ligne par ligne. C’est pourquoi il est essentiel de pouvoir boucler sur les lignes, en utilisant les boucles DO et DO WHILE. On souhaite par exemple créer une table de 100 observations, et une variable aléatoire suivant une loi normale centrée réduite. Avec l’étape DATA :
DATA normale;
DO i=1 TO 100;
x = rannor(0);
OUTPUT;
END;
RUN;Ceci va créer une table contenant la colonne i, soit le numéro de l’observation, et la varible x qui suit la loi normale centrée réduite.
Avec R, pas besoin de boucler. Il est d’ailleurs recommandé de ne pas utiliser les boucles dans R, et si vous pensez “vecteur”, vous n’en aurez quasiment jamais besoin. Pour créer la même table :
normale <- data.frame(i = 1:100, x = rnorm(n = 100, mean = 0, sd = 1))Note : pour voir d’autres fonctions utiles à la création de variables, c’est dans “SAS2R – Episode 2 : Opérateurs et fonctions de base”
Fusion et concaténation de tables
Concaténation
La concaténation de tables consiste à :
- ajouter les lignes de tab2 à tab1
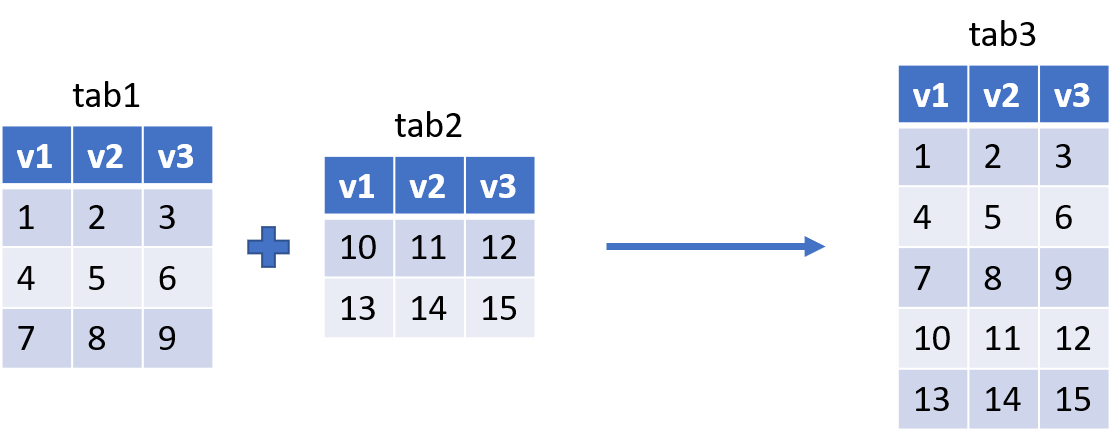
- ou ajouter les colonnes de tab2 à tab1

Dans le cas de la concaténation verticale, c’est-à-dire par lignes, on utilise l’instruction SET de l’étape DATA :
DATA tab3;
SET tab1 tab2;
RUN;Dans R, c’est la fonction rbind() qui fait le job (attention, le nombre et les noms des colonnes doivent être identiques) :
tab3 <- rbind(tab1, tab2)On souhaite parfois garder une trace de l’origine de la donnée en créant une nouvelle colonne. La paramètre IN de l’instruction SET permet cette opération :
DATA tab3;
SET tab1 (in=x) tab2;
IF x=1 THEN origine=”table1”;
ELSE origine=”table2”;
RUN; En R, il faudra au préalable créer une variable ‘origine’ dans chaque table avant de les concaténer :
tab1$origine <- "table1"
tab2$origine <- "table2"
teb3 <- rbind(tab1, tab2)Si on est dans le cas où tab1 et tab2 n’ont pas le même nombre de colonnes et/ou des noms de colonnes différents, la concaténation des deux tables est possible grâce à la fonction bind_rows() du package {dplyr} :
library(dplyr)
tab3 <- tab1 %>% bind_rows(tab2)La concaténation horizontale quand à elle est faisable dans SAS, grâce à l’instruction MERGE. Elle suppose que les colonnes n’ont pas les mêmes noms.
DATA tabC;
MERGE tabA tabB;
RUN;R nous permet également de le faire grâce à la fonction cbind(), lorsque les deux tables présentent le même nombre de lignes. Il n’est pas obligatoire d’avoir une colonne commune comme lors de la fusion de deux tables ; la concaténation horizontale consiste simplement à “coller” deux tables :
tabC <- cbind(tabA, tabB)Avec {dplyr}, vous pourrez utiliser bind_cols() :
tabC <- tabA %>% bind_cols(tabB)La fusion des tables
Avant de faire une jointure entre deux tables, il est impératif de déterminer ce que l’on souhaite garder de chacune d’elles. Il existe en effet plusieurs types de jointures que nous allons développer ci-dessous. La PROC SQL étant plus efficiente et plus complète que l’étape DATA en SAS, nous nous concentrerons sur celle-ci uniquement.
- La jointure INNER JOIN : elle correspond à l’intersection des deux tables.
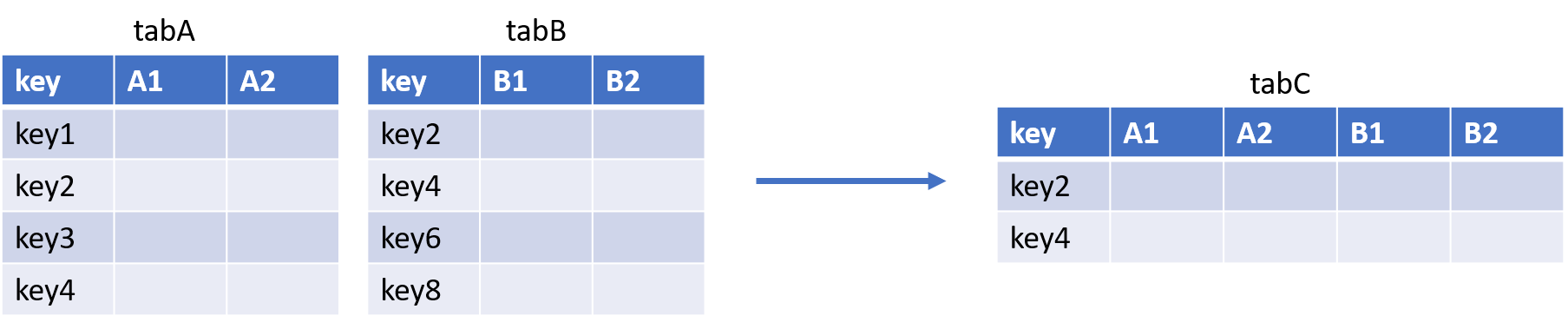
Avec la PROC SQL :
PROC SQL;
SELECT * AS tabC
FROM tabA
INNER JOIN tabB
ON tabA.key = tabB.key;
QUIT;En R, plusieurs fonctions permettent de faire cette jointure. En r-base, ce sera la fonction merge()
tabC <- merge(tabA, tabB, by = "key", all.x = FALSE, all.y = FALSE)Si la clé de jointure est identique pour les deux tables, “key” par exemple, on se contentera d’écrire : by = "key", dans le cas contraire, les paramètres by.x et by.y spécifient le nom de la clé de jointure de chaque table. all.x et all.y représentent les conditions de jointure des tables : avec all.x = TRUE, toutes les lignes de la première table seront conservées, et ceci indépendemment de la correspondance des clés entre les deux tables. Ici, avec all.x = TRUE et all.y = TRUE, seules sont conservées les lignes pour lesquelles la valeur de la clé est présente dans les deux tables.
Le paramètre suffixes permet d’ajouter un suffixe aux noms des colonnes des tables initiales dans la table résultante de la jointure. Par exemple, je souhaite que les colonnes issues de ma première table soient suffixées par « _fromx » et de la seconde par « _fromy » :
tabC <- merge(tabA, tabB, by = "key", all.x = FALSE, all.y = FALSE, suffixes = c("_fromx", "_fromy"))Il est également possible d’utiliser la fonction inner_join() du package {dplyr} :
tabC <- tabA %>% inner_join(tabB, suffix = c("_fromx", "_fromy"))Dans le cas où les clés sont différentes, le paramètre by permet de spécifier les clés de chaque table : by = c("keyA" = "keyB"), et suffix correspond au paramètre suffixes de la fonction merge().
- La jointure LEFT JOIN :
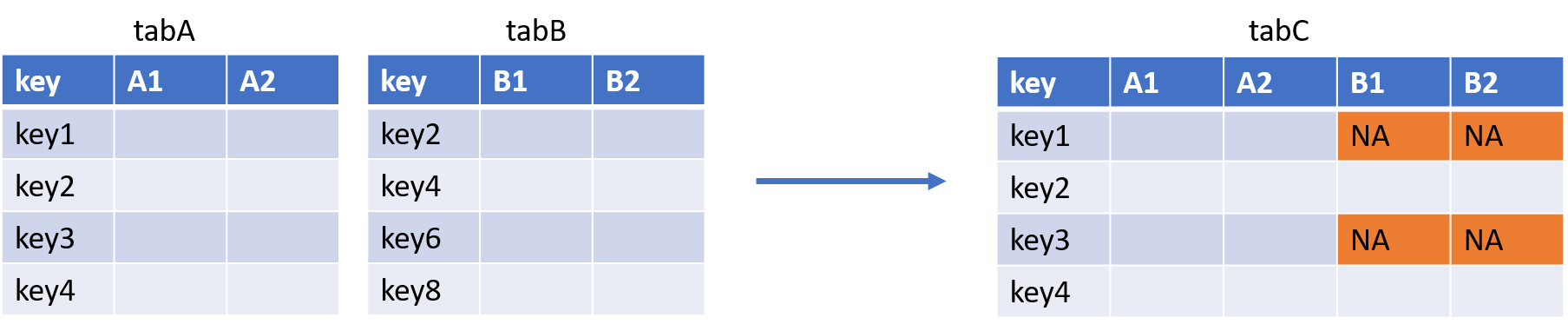
Avec la PROC SQL :
PROC SQL;
SELECT * AS tabC
FROM tabA
LEFT JOIN tabB
ON tabA.key = tabB.key;
QUIT;Avec la fonction merge() :
tabC <- merge(tabA, tabB, by = "key", all.x = TRUE, all.y = FALSE)Puisqu’ici on souhaite garder tous les éléments de la première table, on met le paramètre all.x à TRUE.
Avec la fonction left_join() du package {dplyr}, on s’y retrouve :
tabC <- tabA %>% left_join(tabB)- La jointure RIGHT JOIN :
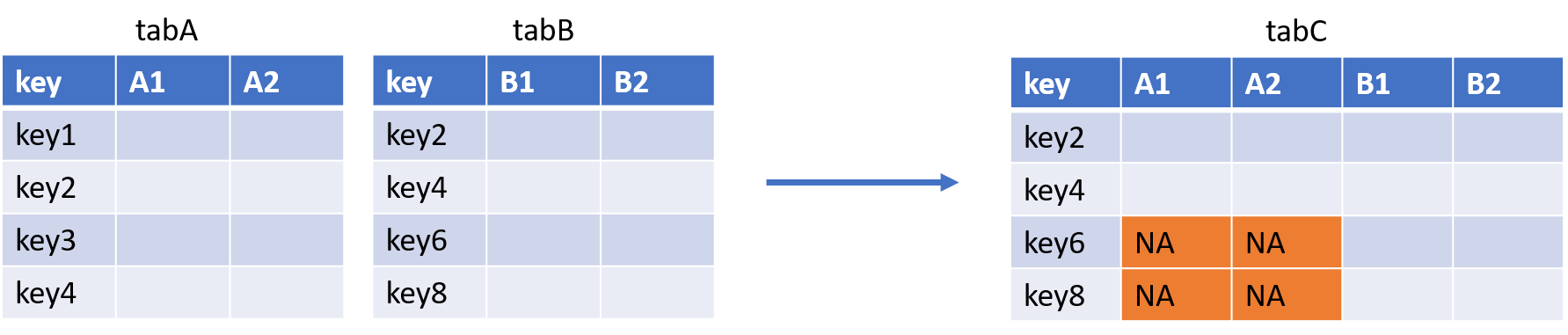
Avec la PROC SQL :
PROC SQL;
SELECT * AS tabC
FROM tabA
RIGHT JOIN tabB
ON tabA.key = tabB.key;
QUIT;Avec la fonction merge() :
tabC <- merge(tabA, tabB, by = "key", all.x = FALSE, all.y = TRUE)Puisqu’ici on souhaite garder tous les éléments de la deuxième table, on met le paramètre all.y à TRUE.
Avec la fonction right_join() du package {dplyr} :
tabC <- tabA %>% right_join(tabB)- La jointure FULL JOIN :
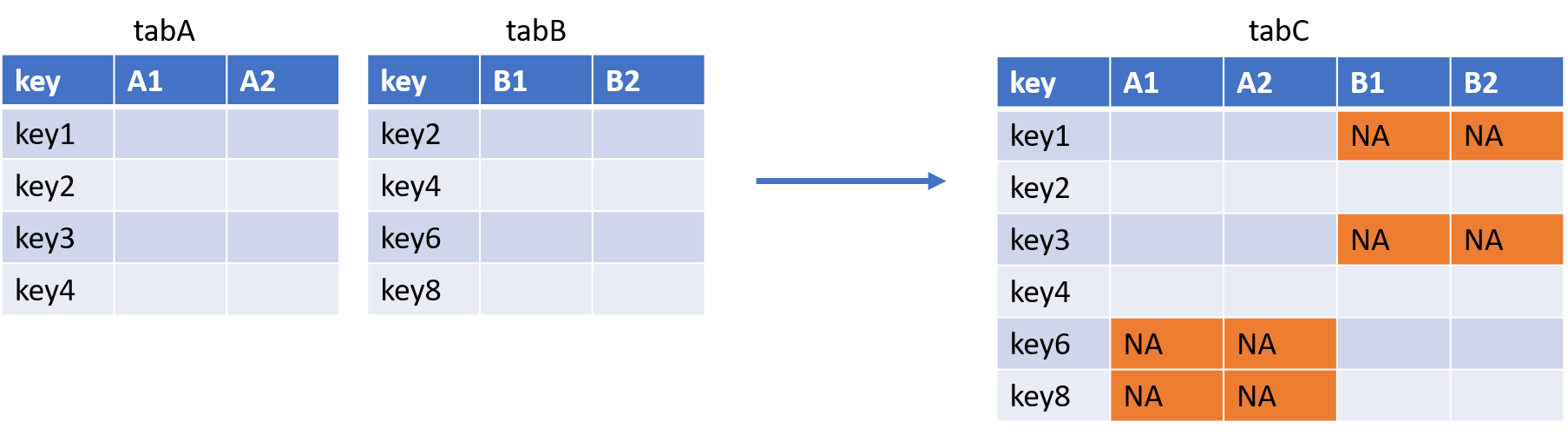
Avec la PROC SQL :
PROC SQL;
SELECT * AS tabC
FROM tabA
FULL JOIN tabB
ON tabA.key = tabB.key;
QUIT;Avec la fonction merge() :
tabC <- merge(tabA, tabB, by = "key", all.x = TRUE, all.y = TRUE)Puisqu’ici on souhaite garder tous les éléments des deux tables, on met les paramètres all.x et all.y à TRUE.
Avec la fonction full_join() du package {dplyr} :
tabC <- tabA %>% full_join(tabB)Note : contrairement à SAS, R ne nécessite pas que les données soient triées avant de faire une jointure.
Dans le prochain épisode…
Ouf ! Je crois qu’on en a fini avec l’étape DATA 🙂 On a bien bossé ! Si vous souhaitez en savoir plus sur l’utilisation de SQL dans R, un peu de patience, on y reviendra bientôt à l’occasion d’un prochain article. Pour le prochain épisode de notre série, nous nous attarderons sur la PROC TRANSPOSE. See you soon !
Dans la même série :




