Tourner, retourner, étirer, pivoter, transposer … Mais quel casse-tête ces données ! Le nom d’une partie de mes colonnes constitue en fait les valeurs d’une certaine variable, cette autre colonne représente en réalité plusieurs variables, et puis chaque ligne contient l’information de deux individus statistiques distincts, et pffff ! J’ai comme envie de retourner sous la couette. Mais je viens de relire la tip top cheat sheet sur l’import des données avec {tidyr} (que vous pouvez retrouver ici : https://readr.tidyverse.org/) qui me redonne de la motivation. Allez au boulot ! Voyons quels genres de puzzles nous attendent !
Note : A l’heure où j’écris cet article, la cheat sheet n’est pas encore mise à jour. Les fonctions qui nous intéressent ici sont gather() et spread() : elles n’ont plus vocation à être utilisées et ont été remplacées par les fonctions pivot_longer() et pivot_wider(), beaucoup plus intuitives, s’inspirant des fonctions melt(), dcast()/ cast() des packages {data.table} et {reshape2}.
Installation et chargement du package :
install.packages("tidyr")
library(tidyr)Sommaire
La notion d’individu statistique
Avant toute chose, il est ici primordial de déterminer en amont nos individus statistiques, c’est-à-dire les observations pour lesquelles on a des mesures qui seront nos variables.
Allonger sa table avec pivot_longer()
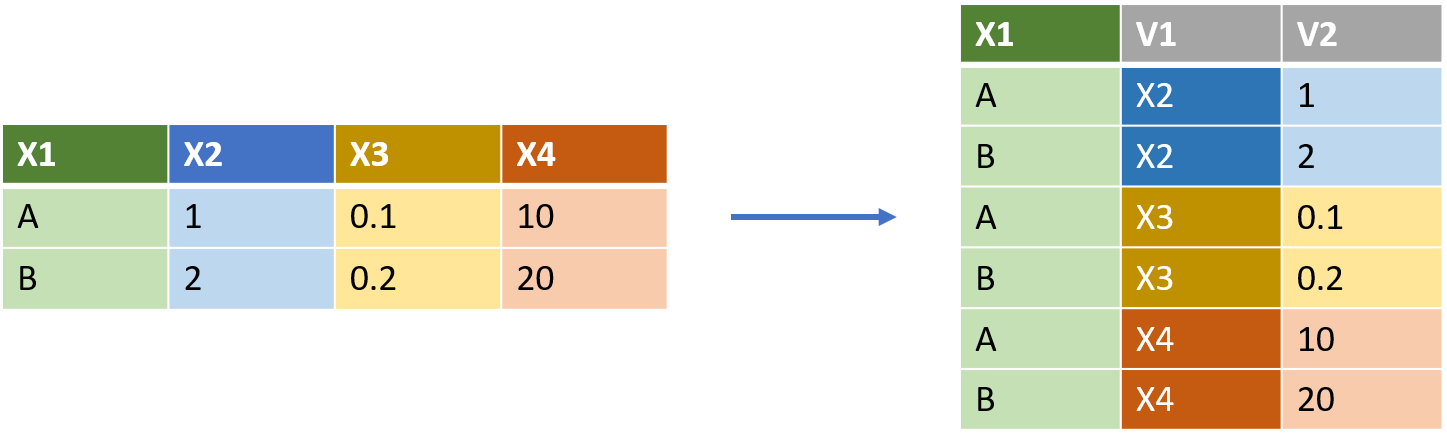
pivot_longer() permet d’allonger ses données en augmentant le nombre de lignes et en diminuant le nombre de colonnes. Ci-dessous, un certain nombre d’exemple de cas que nous pouvons rencontrer.
Des données dans les noms de colonnes
Prenons cet exemple, tiré de la vignette de {data.table} (https://cran.r-project.org/web/packages/data.table/vignettes/datatable-reshape.html) :
family <- data.frame(
family_id = 1:5,
age_mother = c(30, 27, 26, 32, 29),
birth_child1 = c("1998-11-26", "1996-06-22", "2002-07-11", "2004-10-10", "2000-12-05"),
birth_child2 = c("2000-01-29", NA, "2004-04-05", "2009-08-27", "2005-02-28"),
birth_child3 = c(NA, NA, "2007-09-02", "2012-07-21", NA)
)
family## family_id age_mother birth_child1 birth_child2 birth_child3
## 1 1 30 1998-11-26 2000-01-29 <NA>
## 2 2 27 1996-06-22 <NA> <NA>
## 3 3 26 2002-07-11 2004-04-05 2007-09-02
## 4 4 32 2004-10-10 2009-08-27 2012-07-21
## 5 5 29 2000-12-05 2005-02-28 <NA>family_long <- family %>%
pivot_longer(
cols = paste0("birth_child", 1:3), names_to = "child_number",
names_prefix = "birth_child", values_to = "date_of_birth",
values_drop_na = TRUE)
family_long## # A tibble: 11 x 4
## family_id age_mother child_number date_of_birth
## <int> <dbl> <chr> <fct>
## 1 1 30 1 1998-11-26
## 2 1 30 2 2000-01-29
## 3 2 27 1 1996-06-22
## 4 3 26 1 2002-07-11
## 5 3 26 2 2004-04-05
## 6 3 26 3 2007-09-02
## 7 4 32 1 2004-10-10
## 8 4 32 2 2009-08-27
## 9 4 32 3 2012-07-21
## 10 5 29 1 2000-12-05
## 11 5 29 2 2005-02-28Le paramètre cols permet de spécifier les colonnes concernées, c’est-à-dire celles qui contiennent des données. Les paramètres names_to et value_to indiquent les noms des nouvelles colonnes. Le paramètre names_prefix permet de supprimer un préfixe potentiellement présent et inutile dans nos valeurs. Enfin, le paramètre values_drop_na mis à TRUE supprime les lignes pour lesquelles la valeur vaut NA.
Plusieurs variables dans les noms de colonnes
Considérons les données iris :
head(iris)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosaOn a ici en réalité 4 variables : Species, Type qui prendra les valeurs Sepal et Petal, Measure qui prendra les valeurs Length et Width et Value la valeur mesurée. Notre observation devient la longueur (en cm) d’une pièce florale (le sépale ou le pétale) d’une variété d’iris donnée.
On peut alors transformer les données de la manière qui suit :
iris_long <- iris %>%
pivot_longer(
cols = -Species, names_to = c("Type", "Measure"),
names_sep = "[^[:alnum:]]+", values_to = "Value")
iris_long## # A tibble: 600 x 4
## Species Type Measure Value
## <fct> <chr> <chr> <dbl>
## 1 setosa Sepal Length 5.1
## 2 setosa Sepal Width 3.5
## 3 setosa Petal Length 1.4
## 4 setosa Petal Width 0.2
## 5 setosa Sepal Length 4.9
## 6 setosa Sepal Width 3
## 7 setosa Petal Length 1.4
## 8 setosa Petal Width 0.2
## 9 setosa Sepal Length 4.7
## 10 setosa Sepal Width 3.2
## # … with 590 more rowsOn crée 2 variables, le paramètre names_to prend donc comme valeur un vecteur contenant le nom de chacune des deux variables. Il est aussi nécessaire de spécifier le pattern du nom des variables au sein du nom des colonnes, ou encore comme ici ce qui sépare nos deux noms de variables, grâce au paramètre names_sep.
Pour que ce jeu de données soit sans implicites, car c’est bien l’objectif des pivots que d’expliciter et de révéler des variables, il faudrait expliciter l’unité de mesure de nos observations : comment procéderiez-vous ? 😉 Indice : la réponse se trouve sûrement dans notre article « Utiliser la grammaire {dplyr} pour triturer ses données ».
Plusieurs observations par ligne
La difficulté du puzzle augmente d’un cran ! On reconnaît ce type de problématique lorsque l’on observe que les noms de variables se retrouvent dans plusieurs noms de colonnes. Reprenons le premier exemple, en ajoutant une information supplémentaire :
family2 <- data.frame(
family_id = 1:5, age_mother = c(30, 27, 26, 32, 29),
birth_child1 = c("1998-11-26", "1996-06-22", "2002-07-11", "2004-10-10", "2000-12-05"),
birth_child2 = c("2000-01-29", NA, "2004-04-05", "2009-08-27", "2005-02-28"),
gender_child1 = c("M", "M", "F", "M", "F"),
gender_child2 = c("F", NA, "M", "F", "F"))
family2## family_id age_mother birth_child1 birth_child2 gender_child1 gender_child2
## 1 1 30 1998-11-26 2000-01-29 M F
## 2 2 27 1996-06-22 <NA> M <NA>
## 3 3 26 2002-07-11 2004-04-05 F M
## 4 4 32 2004-10-10 2009-08-27 M F
## 5 5 29 2000-12-05 2005-02-28 F FIci, on souhaite donc obtenir les trois variables child_number, birth et gender. Une observation devient l’âge lors de la naissance, la parité ET le sexe d’un enfant pour une mère donnée. Pour ce faire :
family2_long <- family2 %>%
pivot_longer(
cols = -c("family_id", "age_mother"),
names_to = c(".value", "child_number"),
names_sep = "_",
values_drop_na = TRUE)
family2_long## # A tibble: 9 x 5
## family_id age_mother child_number birth gender
## <int> <dbl> <chr> <fct> <fct>
## 1 1 30 child1 1998-11-26 M
## 2 1 30 child2 2000-01-29 F
## 3 2 27 child1 1996-06-22 M
## 4 3 26 child1 2002-07-11 F
## 5 3 26 child2 2004-04-05 M
## 6 4 32 child1 2004-10-10 M
## 7 4 32 child2 2009-08-27 F
## 8 5 29 child1 2000-12-05 F
## 9 5 29 child2 2005-02-28 FIci aussi, on remarque que puisque nous créons plusieurs variables, le paramètre names_to prend donc comme valeur un vecteur, et on spécifie le séparateur de noms de variables grâce au paramètre names_sep. Par ailleurs, .value indique ici que cette partie du nom de la colonne spécifie le nom de la nouvelle variable créée.
Elargir sa table avec pivot_wider()
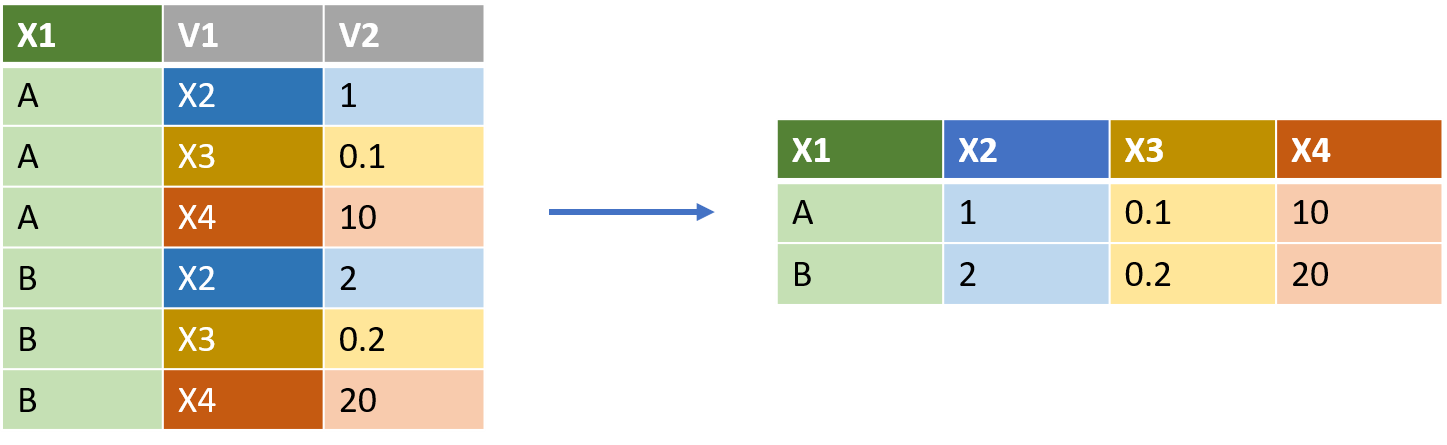
pivot_wider() permet d’élargir ses données en augmentant le nombre de colonnes et en diminuant le nombre de lignes. Il faut bien comprendre que chaque fois qu’on élargit un tableau, de l’information (portée par un nom de variable ou un calcul) devient implicite. Ci-dessous, un certain nombre d’exemples de cas que nous pouvons rencontrer.
Les valeurs d’une variable sont en fait des noms de variables
Prenons l’exemple suivant : je souhaite créer une table de contingence des variables Label et Soil du jeu de données wine du package {FactoMineR}. À l’intersection d’une ligne et d’une colonne : une somme.
library(FactoMineR)
data(wine)
head(wine[,c("Label", "Soil")])## Label Soil
## 2EL Saumur Env1
## 1CHA Saumur Env1
## 1FON Bourgueuil Env1
## 1VAU Chinon Env2
## 1DAM Saumur Reference
## 2BOU Bourgueuil Reference# ce que je souhaite obtenir, sous forme de data.frame :
table(wine$Label, wine$Soil)##
## Reference Env1 Env2 Env4
## Saumur 3 3 3 2
## Bourgueuil 3 3 0 0
## Chinon 1 1 2 0# ce que j'obtiens :
df_table <- as.data.frame(table(wine$Label, wine$Soil)) %>%
setNames(c("Label", "Soil", "Freq"))
df_table## Label Soil Freq
## 1 Saumur Reference 3
## 2 Bourgueuil Reference 3
## 3 Chinon Reference 1
## 4 Saumur Env1 3
## 5 Bourgueuil Env1 3
## 6 Chinon Env1 1
## 7 Saumur Env2 3
## 8 Bourgueuil Env2 0
## 9 Chinon Env2 2
## 10 Saumur Env4 2
## 11 Bourgueuil Env4 0
## 12 Chinon Env4 0# mise en forme grâce à pivot_wider() :
df_table_wider <- df_table %>%
pivot_wider(names_from = Soil, values_from = Freq)
df_table_wider## # A tibble: 3 x 5
## Label Reference Env1 Env2 Env4
## <fct> <int> <int> <int> <int>
## 1 Saumur 3 3 3 2
## 2 Bourgueuil 3 3 0 0
## 3 Chinon 1 1 2 0Bon, vous me direz que j’aurais très bien pu faire ça à la place : as.data.frame.matrix(table(wine$Label, wine$Soil)), mais c’était pour illustrer mon propos et économiser de la frappe de parenthèse alors vous ne m’en voudrez pas 😉
Le paramètre names_from indique la variable pour laquelle les valeurs représentent des variables, et values_from la variable contenant les valeurs prises par ces variables.
Agrégation
Continuons avec nos données de vin. Je souhaite maintenant connaître la valeur moyenne de la variable Overall.quality par Label et par Soil. La fonction pivot_wider() nous permet cela :
wine_means <- wine %>%
as_tibble() %>%
dplyr::select(Label, Soil, Overall.quality) %>%
pivot_wider(
names_from = Soil,
values_from = Overall.quality,
values_fn = list(Overall.quality = mean))
wine_means## # A tibble: 3 x 5
## Label Env1 Env2 Reference Env4
## <fct> <dbl> <dbl> <dbl> <dbl>
## 1 Saumur 3.33 3.30 3.75 2.75
## 2 Bourgueuil 3.18 NA 3.70 NA
## 3 Chinon 3.2 2.96 3.54 NALe paramètre values_fn permet d’indiquer quelle fonction appliquer à la variable que l’on souhaite agréger (ici, la moyenne).
Générer des noms de colonne à partir de plusieurs variables
Imaginons le jeu de données suivant : nous avons pour 3 cépages différents et provenant de 2 types d’environnements l’évolution de leur note globale entre 2010 et 2015 :
data_vin <- expand.grid(
cepage = c("Malbec", "Cabernet", "Syrah"),
env = c("env1", "env2"),
annee = 2010:2015
) %>%
dplyr::mutate(note = runif(nrow(.), 0, 5))
data_vin## # A tibble: 36 x 4
## cepage env annee note
## <chr> <chr> <int> <dbl>
## 1 Malbec env1 2010 2.96
## 2 Malbec env1 2011 1.86
## 3 Malbec env1 2012 0.123
## 4 Malbec env1 2013 4.38
## 5 Malbec env1 2014 3.96
## 6 Malbec env1 2015 3.35
## 7 Malbec env2 2010 4.84
## 8 Malbec env2 2011 1.65
## 9 Malbec env2 2012 4.26
## 10 Malbec env2 2013 2.37
## # … with 26 more rowsOn souhaite maintenant obtenir un jeu de données ayant pour colonne chaque combinaison cépage/environnement. Il suffit d’attribuer au paramètre names_from un vecteur contenant le nom des deux variables :
data_vin %>%
pivot_wider(
names_from = c(cepage, env),
values_from = note
)## # A tibble: 6 x 7
## annee Malbec_env1 Malbec_env2 Cabernet_env1 Cabernet_env2 Syrah_env1 Syrah_env2
## <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2010 2.96 4.84 4.75 1.90 1.78 3.78
## 2 2011 1.86 1.65 2.61 2.04 1.84 3.52
## 3 2012 0.123 4.26 4.93 3.40 4.50 0.687
## 4 2013 4.38 2.37 3.50 4.22 3.41 2.75
## 5 2014 3.96 3.56 0.533 1.26 0.628 0.912
## 6 2015 3.35 2.93 1.93 3.84 3.60 2.78Pour conclure… et aller plus loin !
On l’a vu, les nouvelles fonctions pivot_longer() et pivot_wider() du package {tidyr} ont été refondues pour faciliter leur utilisation et les noms des paramètres sont plus parlants, intuitifs que leurs ancêtres gather() and spread(). C’est l’esprit du {tidyverse}. Ces fonctions permettent de pivoter un large spectre de jeux de données, mais il est toutefois possible que pour des puzzles plus complexes elles ne soient pas suffisantes. Pour les problèmes de pivot plus élaborés, vous pourrez faire appel aux fonctions build_longer_spec()/pivot_longer_spec() et build_wider_spec()/pivot_wider_spec().
Pour plus d’exemples et plus d’explications sur les fonctions *_spec(), je vous invite à consulter cet article dédié au pivot (en anglais): https://tidyr.tidyverse.org/articles/pivot.html





Laisser un commentaire